Нурк С. та ін. Повна послідовність геному людини. Наука 37644–53 (2022).
McCarthy, S. та ін. Еталонна панель із 64 976 гаплотипів для визначення генотипу. Нац. Жене. 481279–1283 (2016).
Menschaert, G. & Fenyö, D. Протеогеноміка з точки зору біоінформатики: поле, що розвивається. Мас-спектр. Рев. 36584–599 (2017).
Несвіжський А. І. Протеогеноміка: концепції, застосування та обчислювальні стратегії. Нац. методи 111114–1125 (2014).
Wang, X. & Zhang, B. customProDB: пакет R для створення налаштованих баз даних білка з даних RNA-Seq для пошуку в протеоміці. Біоінформатика 293235–3237 (2013).
Umer, HM та ін. Створення баз даних протеогеноміки на основі ENSEMBL сприяє ідентифікації неканонічних пептидів. Біоінформатика 381470–1472 (2022).
Spooner, W. та ін. Гаплозавр обчислює білкові гаплотипи для використання в розробці точних ліків. Нац. Поширений. 94128 (2018).
Cao, X. & Xing, J. PrecisionProDB: покращення продуктивності протеоміки для прецизійної медицини. Біоінформатика 373361–3363 (2021).
Ліцензія MIT (2024) https://choosealicense.com/licenses/mit/ (перевірено 24 січня 2024).
Auton, A. та ін. Глобальна довідка про генетичні варіації людини. природа 52668–74 (2015).
Lowy-Gallego, E. та ін. Варіант із використанням збірки GRCh38 із даними третьої фази проекту 1000 геномів. Welcome Open Res 450 (2019).
Vašíček, J. Послідовності білкових гаплотипів, отримані ProHap із набору даних 1000 Genomes Project. Зенодо https://zenodo.org/records/12671237 (2024).
Vašíček, J. Послідовності білкових гаплотипів, отримані ProHap з набору даних Haplotype Reference Consortium Release 1.1. Зенодо https://doi.org/10.5281/zenodo.12671302 (2024).
Ляо, В.-В. та ін. Проект посилання на пангеном людини. природа 617312–324 (2023).
Vašíček, J. Послідовності білкових гаплотипів, отримані ProHap з набору даних Human Pangenome Reference Consortium. Зенодо https://doi.org/10.5281/zenodo.12686819 (2024).
Vašíček, J. та ін. Знаходження гаплотипових ознак у білках. GigaScience 12giad093 (2023).
Geyer, PE та ін. Протеоміка виявляє вплив тривалої втрати ваги на протеом плазми людини. мол. сист. Biol. 12901 (2016).
Vašíček, J. & Skiadopoulou, D. Повторна обробка набору даних «Профілювання протеома плазми виявляє вплив втрати ваги на сімейство аполіпопротеїнів і статус системного запалення». Зенодо https://doi.org/10.5281/zenodo.12725746 (2024).
Бадер, Дж. М., Альбрехт, В. і Манн, М. Протеоміка рідин організму на основі MS: кінець початку. мол. Стільниковий. Протеоміка 22100577 (2023).
Skiadopoulou, D. та ін. Час утримання та предиктори фрагментації підвищують впевненість у ідентифікації поширених варіантів пептидів. J. Proteome Res. 223190–3199 (2023).
Морено-Естрада, А. та ін. Генетика Мексики повторює субструктуру індіанців і впливає на біомедичні ознаки. Наука 3441280–1285 (2014).
Фокс, К. Ілюзія інклюзії: дослідницька програма «Усі ми» та ДНК корінних народів. Н. англ. J. Med. 383411–413 (2020).
Hudson, M. та ін. Права, інтереси та очікування: погляди корінного населення на необмежений доступ до геномних даних. Нац. Преподобний Женет. 21377–384 (2020).
Birney, E., Inouye, M., Raff, J., Rutherford, A. & Scally, A. Мова раси, етнічної приналежності та походження в генетичних дослідженнях людини. Препринт на https://doi.org/10.48550/arXiv.2106.10041 (2021).
Cunningham, F. та ін. Ансамбль 2022. Nucleic Acids Res. 50D988–D995 (2022).
Kowalski, MH та ін. Використання >100 000 послідовностей цілого генома консорціуму NHLBI Trans-Omics for Precision Medicine (TOPMed) покращує якість імпутації та виявлення асоціацій рідкісних варіантів у змішаному африканському та латиноамериканському/латиноамериканському населеннях. PLoS Genet. 15e1008500 (2019).
Morales, J. та ін. Спільний набір стенограм NCBI та EMBL-EBI для клінічної геноміки та досліджень. природа 604310–315 (2022).
Declercq, A. та ін. РС2Rescore: перерахунок на основі даних значно підвищує швидкість ідентифікації імунопептидів. мол. Стільниковий. Протеоміка 21(8), 100266 (2022).
Hickey, G. та ін. Побудова графа пангенома з вирівнювання генома за допомогою Minigraph-Cactus. Нац. Біотехнологія. 42663–673 (2024).
Stawiński, P. & Płoski, R. Genebe.net: впровадження та підтвердження автоматичного призначення критеріїв патогенності варіанту ACMG. Clin. Жене. 106119–126 (2024).
Водел, М., Барснес, Х., Бервен, Ф. С., Сікманн, А. та Мартенс, Л. SearchGUI: графічний інтерфейс користувача з відкритим кодом для одночасного пошуку OMSSA та X!Tandem. Протеоміка 11996–999 (2011).
Водель, М. та ін. PeptideShaker дає змогу повторно аналізувати набори протеомних даних, отриманих із MS. Нац. Біотехнологія. 3322–24 (2015).
Fenyö, D. & Beavis, RC Метод оцінки статистичної значущості ідентифікації білка на основі мас-спектрометрії з використанням загальних схем оцінки. анальний Chem. 75768–774 (2003).
Park, CY, Klammer, AA, Käll, L., MacCoss, MJ & Noble, WS. Швидка та точна ідентифікація пептиду з тандемних мас-спектрів. J. Proteome Res. 73022–3027 (2008).
Käll, L., Canterbury, JD, Weston, J., Noble, WS & MacCoss, MJ. Напівкероване навчання для ідентифікації пептидів із наборів даних протеоміки дробовика. Нац. методи 4923–925 (2007).
Bouwmeester, R., Gabriels, R., Hulstaert, N., Martens, L. & Degroeve, S. DeepLC може передбачити час утримування для пептидів, які несуть ще невідомі модифікації. Нац. методи 181363–1369 (2021).
Declercq, A. та ін. Оновлений веб-сервер MS2PIP підтримує найсучасніші програми протеоміки. Nucleic Acids Res. 51W338–W342 (2023).
Unger, L., Mathisen, AF, Chera, S., Legøy, TA & Ghila, L. Код GLI контролює рівні HNF1A під час диференціації передньої кишки. Міжн. J. Dev. Biol. https://doi.org/10.1387/ijdb.230220lg (2024).
Конг, А. Т., Лепревост, Ф. В., Автономов, Д. М., Меллахеруву, Д. і Несвіжський, А. І. MSFragger: надшвидка та повна ідентифікація пептидів у протеоміці на основі мас-спектрометрії. Нац. методи 14513–520 (2017).
Teo , GC , Polasky , DA , Yu , F. & Nesvizhskii , AI Алгоритм швидкого деізотопування та його реалізація в пошуковій системі MSFragger . J. Proteome Res. 20498–505 (2021).
Yang, KL та ін. MSBooster: покращує показники ідентифікації пептидів за допомогою функцій глибокого навчання. Нац. Поширений. 144539 (2023).
Claeys, T. та ін. lesSDRF — це більше: максимізація цінності даних протеоміки за допомогою спрощеної анотації метаданих. Нац. Поширений. 146743 (2023).
Käll, L., Storey, JD & Noble, WS. Непараметрична оцінка ймовірностей задньої помилки, пов’язаної з пептидами, визначеними тандемною мас-спектрометрією. Біоінформатика 24i42–i48 (2008).
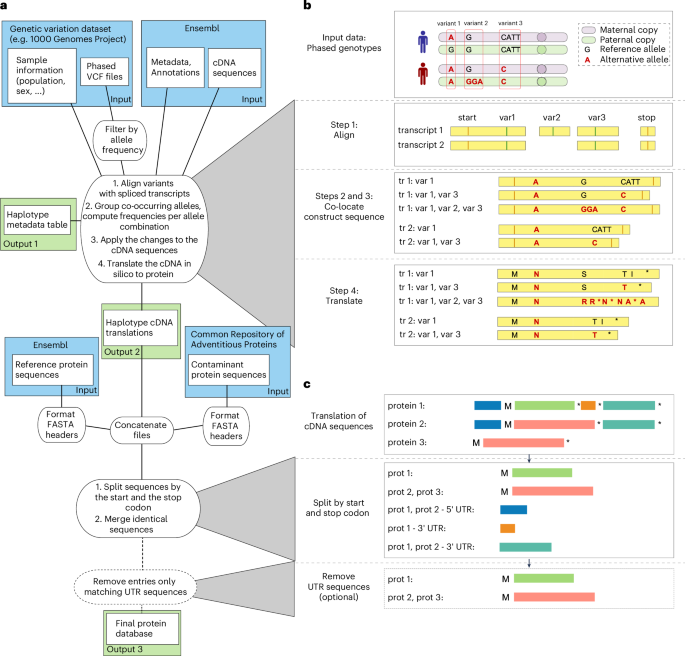
Залишити відповідь